状態空間モデル - R dlm (1)
状態空間モデル(正確には、時系列モデルの状態空間表現)の学習メモ。Rのdlmパッケージを使う。
参考書は ”和合 2013”。
単純なモデル(ローカルレベルモデル)を状態空間表現し、カルマンフィルタを実行してみる。
動的線形モデル(Dynamic Linear Model) / 線形ガウス状態空間モデル
t=0, θ[0]~N(m[0], C[0])
t>0, Y[t] = F[t] θ[t] + v[t], v[t]~N(0, V[t]) 観測値
θ[t] = G[t]θ[t-1] + w[t], w[t]~N(0, W[t]) 状態
Yはm次元のベクトル、θはp次元のベクトルとするが、多変量時系列でなければm=1とシンプルになる。
m=1とすると、それぞれの要素の次元は、Fが1×p、v、Vはスカラー、Gはp×p、wはp×1、Wはp×pとなる。
式のパラメータ(F[t]、V[t]、G[t]、W[t])は[t]を付けた時間依存を表す形式で表記したが、多くの場合は時間依存とすることはない。
状態(θ)とは観測不能で、あるシステムを構成する要素、観測値(Y)は状態より発生する現象から実際に観測できるものを解釈する。
カルマンフィルタにより与えられたデータからθとYの分布を推定することができる。
イメージ図。
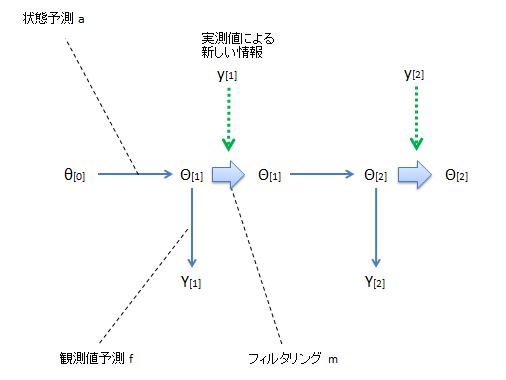
θ[0]から始まり、θ[1]を予測し、Y[1]を予測する。そして新たなデータy[1]が観測され、その情報を用いてθ[1]が修正される(フィルタリング)。
そのθ[1]からθ[2]を予測し、Y[2]を予測する。そして新たなデータy[2]が観測され、その情報を用いてθ[2]が修正される
これを繰り返しにより、θ、Yの分布が求められる。
各予測値とフィルタリング値の分布は以下に定義される。動的線形モデルは正規分布を仮定しているので、平均と分散により分布が特定される。
t=0時点の状態の分布、θ[0]~N(m[0], C[0])、が与えられているとする。
(1) Y[1:t-1](Y[1],Y[2], … , Y[t-1])が与えられた元での1期先状態予測分布
θ[t]|Y[1:t-1] ~ N(a[t], R[t])
a[t] = G[t] m[t-1]
R[t] = G[t] C[t-1] G[t]’ + W[t]
(2) Y[1:t-1]が与えられた元での1期先観測予測分布
Y[t]|Y[1:t-1] ~ N(f[t], Q[t])
f[t] = F[t] a[t]
Q[t] = F[t] R[t] F[t]’ + V[t]
(3) Y[1:t]が与えられた元でのフィルタリング分布
θ[t]|Y[1:t] ~ N(m[t], C[t])
m[t] = a[t] + R[t] F[t]’ Q[t]^-1 e[t] e[t] = Y[t] – f[t]
C[t] = R[t] - R[t] F[t]’ Q[t]^-1 F[t] R[t]
カルマンフィルタは基本的にY[1:t]が与えられたときのθ[t]のフィルタリング分布の推定を目的としている。Y[1:t]が与えられたときのθ[t+k]やY[t+k]のk期先予測の分布に関しては後述(k>1)。
この分野でもっともシンプルなモデルとして用いられるローカルレベルモデルで、状態空間モデルの定義やカルマンフィルタを試してみる。
ローカルレベルモデル (ランダムウォーク+ノイズモデル)では、F=1、G=1と定義される。
t=0, θ[0]~N(m[0], C[0])
t>0, Y[t] = θ[t] + v[t], v[t]~N(0, V) 観測値
θ[t] =θ[t-1] + w[t], w[t]~N(0, W) 状態
θ[t]が時刻tでの真の値、Y[t]がθ[t]の観測値、v[t]が観測誤差。θ[t]は一期先の値θ[t-1]にランダムな値w[t]が加わって変化するシステムだと考える単純なモデル。
θをランダムウォークとすると、Yはそれに誤差vを加えたもの。
# m[0]=10、c[0]=5
θ[0]=10、V=3、W=6と設定して、シミュレーションで長さ20データを作成してみる。
library(dlm)
V <- 3
W <- 6
theta.0 <- 10
time <- c(0)
theta <- c(theta.0)
y <- c(NA)
for(i in 1:20){
time <- append(time, i)
theta_next <- theta[i] + rnorm(1, mean=0, sd=sqrt(W))
theta <- append(theta, theta_next)
y_next <- theta_next + rnorm(1, mean=0, sd=sqrt(V))
y <- append(y, y_next)
}
df1 <- data.frame(time=time, theta=theta, y=y)
minY <- min(df1$theta)-10
maxY <-max(df1$theta)+10
plot(df1$time, df1$theta, ylim=c(minY,maxY),type="o", col="blue", ylab="val(theta, y)")
par(new=TRUE)
plot(df1$time, df1$y, ylim=c(minY,maxY), type="o", ylab="")

青:θ、黒:Y
では、ここから逆に、このデータ(Yのみ)が与えられたとして、カルマンフィルタにより状態と観測値の推定を行う。
当てはめの際は、まずモデルを定義する。
モデルは上でデータを作成したとおり、ローカルレベルモデルとし、m[0]=10、C[0]=50と仮定する。また、V=3、W=6は既知とする。
(状態の事前分布N(m[0], C[0])に関する情報が不確かな場合、C[0]を大きく仮定するらしい。)
dlm()関数を使用した場合は以下。
m.0 <- 10
C.0 <- 50
mod1 <- dlm( m0=m.0, C0=C.0, FF=1, V=3, GG=1, W=6 )
unlist(mod1)
m0 C0 FF V GG W 10 50 1 3 1 6
class(mod1) # "dlm"
typeof(mod1) # "list"
dlmModPoly()を使ってもローカルレベルモデルを定義できる。
mod2 <- dlmModPoly( order=1, m0=m.0, C0=C.0, dV=3, dW=6 )
unlist(mod2)
m0 C0 FF V GG W 10 50 1 3 1 6
dlmFilter(データ, 当てはめるモデル)により、カルマンフィルタを実行し、1期先状態予測、1期先観測値予測、フィルタリングの値を計算する。
fit_mod1 <- dlmFilter(df1$y[-1], mod1)
str(fit_mod1, 1)
List of 9
$ y : num [1:20] 11.48 14.89 16.27 15.19 7.64 ...
$ mod:List of 6
..- attr(*, "class")= chr "dlm"
$ m : num [1:21] 10 11.4 14 15.7 15.3 ...
$ U.C:List of 21
$ D.C: num [1:21, 1] 7.07 1.69 1.5 1.48 1.48 ...
$ a : num [1:20] 10 11.4 14 15.7 15.3 ...
$ U.R:List of 20
$ D.R: num [1:20, 1] 7.48 2.97 2.87 2.86 2.86 ...
$ f : num [1:20] 10 11.4 14 15.7 15.3 ...
- attr(*, "class")= chr "dlmFiltered"
fit_mod1$a # 1期先状態予測
fit_mod1$f # 1期先観測値予測
fit_mod1$m # フィルタリング
データと、それら計算結果をまとめたデータフレームを作成。
df_res1 <- data.frame(time=df1$time, y=df1$y, theta=df1$theta, a=c(NA,fit_mod1$a), f=c(NA,fit_mod1$f), m=fit_mod1$m)
time y theta a f m 1 0 NA 10.00000 NA NA 10.000000 2 1 11.480221 11.12716 10.000000 10.000000 11.404956 3 2 14.887411 14.39119 11.404956 11.404956 14.005587 4 3 16.268663 14.81166 14.005587 14.005587 15.664657 5 4 15.192051 14.39024 15.664657 15.664657 15.318650 6 5 7.640275 11.47224 15.318650 15.318650 9.697648 7 6 11.918582 12.24209 9.697648 9.697648 11.323486 8 7 11.739846 13.41268 11.323486 11.323486 11.628283 9 8 19.019994 18.91138 11.628283 11.628283 17.039391 10 9 21.572069 19.41500 17.039391 17.039391 20.357542 11 10 20.391132 17.97181 20.357542 20.357542 20.382131 12 11 15.116908 18.52915 20.382131 20.382131 16.527721 13 12 19.366015 21.61766 16.527721 16.527721 18.605496 14 13 21.751131 19.29930 18.605496 18.605496 20.908260 15 14 16.585866 17.66393 20.908260 20.908260 17.744048 16 15 17.432607 16.43332 17.744048 17.744048 17.516058 17 16 22.007343 19.82772 17.516058 17.516058 20.803907 18 17 18.873734 17.81004 20.803907 20.803907 19.390922 19 18 19.547199 19.00535 19.390922 19.390922 19.505325 20 19 17.828754 19.51896 19.505325 19.505325 18.277990 21 20 23.217935 22.09485 18.277990 18.277990 21.894281
plot(df_res1$time, df_res1$theta, ylim=c(minY,maxY),type="o", col="blue", ylab="val") # 状態
par(new=TRUE)
plot(df_res1$time, df_res1$y, ylim=c(minY,maxY), type="o", ylab="") # 観測値
par(new=TRUE)
plot(df_res1$time, df_res1$a, ylim=c(minY,maxY), type="l", col="green", ylab="") # 1期先状態予測
par(new=TRUE)
plot(df_res1$time, df_res1$f, ylim=c(minY,maxY), type="l", col="red", ylab="") # 1期先観測値予測
par(new=TRUE)
plot(df_res1$time, df_res1$m, ylim=c(minY,maxY), type="l", col="purple", ylab="") # フィルタリング

青(点と線):θ、黒(点と線):Y
緑:1期先状態予測、赤:1期先観測値予測、紫:フィルタリング
ローカルレベルモデルの場合は、1期先状態予測と1期先観測値予測は同じ(a[t] = f[t])となる。よって、図では緑が赤で上書きされている。
また、1期先状態予測は1期前のフィルタリングと同じ(a[t] = m[t-1])となる。
分散を見てみる。
時普遍のモデルの場合、C[t]はtが増えるに従って極値に近づくらしい。
以下の指定で、フィルタリング結果から分散(RとCのみ)を取り出せる。
R <- unlist(dlmSvd2var(fit_mod1$U.R, fit_mod1$D.R))
C <- unlist(dlmSvd2var(fit_mod1$U.C, fit_mod1$D.C))
df_res1 <- data.frame(df_res1, R=c(NA,R), C=C)
time y theta a f m R C 1 0 NA 10.00000 NA NA 10.000000 NA 50.000000 2 1 11.480221 11.12716 10.000000 10.000000 11.404956 56.000000 2.847458 3 2 14.887411 14.39119 11.404956 11.404956 14.005587 8.847458 2.240343 4 3 16.268663 14.81166 14.005587 14.005587 15.664657 8.240343 2.199313 5 4 15.192051 14.39024 15.664657 15.664657 15.318650 8.199313 2.196379 6 5 7.640275 11.47224 15.318650 15.318650 9.697648 8.196379 2.196169 7 6 11.918582 12.24209 9.697648 9.697648 11.323486 8.196169 2.196154 8 7 11.739846 13.41268 11.323486 11.323486 11.628283 8.196154 2.196153 9 8 19.019994 18.91138 11.628283 11.628283 17.039391 8.196153 2.196152 10 9 21.572069 19.41500 17.039391 17.039391 20.357542 8.196152 2.196152 11 10 20.391132 17.97181 20.357542 20.357542 20.382131 8.196152 2.196152 12 11 15.116908 18.52915 20.382131 20.382131 16.527721 8.196152 2.196152 13 12 19.366015 21.61766 16.527721 16.527721 18.605496 8.196152 2.196152 14 13 21.751131 19.29930 18.605496 18.605496 20.908260 8.196152 2.196152 15 14 16.585866 17.66393 20.908260 20.908260 17.744048 8.196152 2.196152 16 15 17.432607 16.43332 17.744048 17.744048 17.516058 8.196152 2.196152 17 16 22.007343 19.82772 17.516058 17.516058 20.803907 8.196152 2.196152 18 17 18.873734 17.81004 20.803907 20.803907 19.390922 8.196152 2.196152 19 18 19.547199 19.00535 19.390922 19.390922 19.505325 8.196152 2.196152 20 19 17.828754 19.51896 19.505325 19.505325 18.277990 8.196152 2.196152 21 20 23.217935 22.09485 18.277990 18.277990 21.894281 8.196152 2.196152
t>=3 くらいからC[t]はほぼ極値となったようである。
状態の1期先予測の分散であるR[t]はY[t]が与えられた後に計算されるC[t]より大きくなる。
k期先予測(k>1)を考える。
Y[1:t]が与えられたときのθ[t+k]やY[t+k]の分布の予測に関しては後述(k>1)。
t時点の状態の分布、θ[t]|Y[t]~N(m[t], C[t])、が与えられているとする。
m[t] = a[t]、C[t] = R[t]と置き換え、カルマンフィルタで見たように、状態と観測値の推定が繰り返される。
(1) Y[1:t]が与えられた元でのk期先状態予測分布
θ[t+k]|Y[1:t] ~ N(a[t+k], R[t+k])
a[t+k] = G[t+k] a[t+k-1]
R[t+k] = G[t+k] R[t+k-1] G[t+k]’ + W[t+k]
k期先の状態の予測分布においては、1期先で見た式と比べると、m、Cがa、Rに置き換わっただけである。要するに、推定されないフィルタリング分布を自身の状態予測分布で置き換え、逐次推定を繰り返している。
(2) Y[1:t]が与えられた元でのk期先観測予測分布
Y[t+k]|Y[1:t] ~ N(f[t+k], Q[t+k])
f[t+k] = F[t+k] a[t+k]
Q[t+k] = F[t+k] R[t+k] F[t+k]’ + V[t+k]
以上が定義される。
dlmForecast(あてはめたモデル, 予測期間)により、k期先状態予測、k期先観測値予測、フィルタリングの値を計算する。
k=10とする。
k <- 10 # k=10
pred_mod1 <- dlmForecast(fit_mod1, nAhead=k)
str(pred_mod1, 1)
List of 4 $ a: num [1:10, 1] 21.9 21.9 21.9 21.9 21.9 ... $ R:List of 10 $ f: num [1:10, 1] 21.9 21.9 21.9 21.9 21.9 ... $ Q:List of 10
df_pred1 <- data.frame(time=21:30, a=pred_mod1$a, R=unlist(pred_mod1$R), f=pred_mod1$f, Q=unlist(pred_mod1$Q))
# df_res1とdf_pred1の必要項目を結合
tm <- c(df_res1$time, df_pred1$time)
tt <- c(df_res1$theta, rep(NA,nrow(df_pred1)))
y <- c(df_res1$y, rep(NA,nrow(df_pred1)))
m <- c(df_res1$m, rep(NA,nrow(df_pred1)))
a <- c(df_res1$a, df_pred1$a)
R <- c(df_res1$R, df_pred1$R)
f <- c(df_res1$f, df_pred1$f)
Q <- c(rep(NA,nrow(df_res1)), df_pred1$Q)
df_all1 <- data.frame(time=tm, theta=tt, y=y, m=m, a=a, R=R, f=f, Q=Q)
time theta y m a R f Q 1 0 10.00000 NA 10.000000 NA NA NA NA 2 1 11.12716 11.480221 11.404956 10.000000 56.000000 10.000000 NA 3 2 14.39119 14.887411 14.005587 11.404956 8.847458 11.404956 NA 4 3 14.81166 16.268663 15.664657 14.005587 8.240343 14.005587 NA 5 4 14.39024 15.192051 15.318650 15.664657 8.199313 15.664657 NA 6 5 11.47224 7.640275 9.697648 15.318650 8.196379 15.318650 NA 7 6 12.24209 11.918582 11.323486 9.697648 8.196169 9.697648 NA 8 7 13.41268 11.739846 11.628283 11.323486 8.196154 11.323486 NA 9 8 18.91138 19.019994 17.039391 11.628283 8.196153 11.628283 NA 10 9 19.41500 21.572069 20.357542 17.039391 8.196152 17.039391 NA 11 10 17.97181 20.391132 20.382131 20.357542 8.196152 20.357542 NA 12 11 18.52915 15.116908 16.527721 20.382131 8.196152 20.382131 NA 13 12 21.61766 19.366015 18.605496 16.527721 8.196152 16.527721 NA 14 13 19.29930 21.751131 20.908260 18.605496 8.196152 18.605496 NA 15 14 17.66393 16.585866 17.744048 20.908260 8.196152 20.908260 NA 16 15 16.43332 17.432607 17.516058 17.744048 8.196152 17.744048 NA 17 16 19.82772 22.007343 20.803907 17.516058 8.196152 17.516058 NA 18 17 17.81004 18.873734 19.390922 20.803907 8.196152 20.803907 NA 19 18 19.00535 19.547199 19.505325 19.390922 8.196152 19.390922 NA 20 19 19.51896 17.828754 18.277990 19.505325 8.196152 19.505325 NA 21 20 22.09485 23.217935 21.894281 18.277990 8.196152 18.277990 NA 22 21 NA NA NA 21.894281 8.196152 21.894281 11.19615 23 22 NA NA NA 21.894281 14.196152 21.894281 17.19615 24 23 NA NA NA 21.894281 20.196152 21.894281 23.19615 25 24 NA NA NA 21.894281 26.196152 21.894281 29.19615 26 25 NA NA NA 21.894281 32.196152 21.894281 35.19615 27 26 NA NA NA 21.894281 38.196152 21.894281 41.19615 28 27 NA NA NA 21.894281 44.196152 21.894281 47.19615 29 28 NA NA NA 21.894281 50.196152 21.894281 53.19615 30 29 NA NA NA 21.894281 56.196152 21.894281 59.19615 31 30 NA NA NA 21.894281 62.196152 21.894281 65.19615
ローカルレベルモデルなので、a[t+k] = f[t+k] = m[t]となる。ランダム項が与えられるのみで構成されるので、一度推定されたm[t]と、k期先状態と観測値予測は同じとなる。
分散は、期間が先になるほど広がって行くのが分かる。
状態と観測値予測をプロットしてみる。
# aとfの95%CI
ci_a <- cbind(df_all1$a+df_all1$R*qnorm(0.025), df_all1$a+df_all1$R*qnorm(0.975))
ci_f <- cbind(df_all1$f+df_all1$Q*qnorm(0.025), df_all1$f+df_all1$Q*qnorm(0.975))
minY <- min(na.omit(df_all1$theta))-30
maxY <-max(na.omit(df_all1$theta))+30
plot(df_all1$time, df_all1$theta, xlim=c(0,30), ylim=c(minY,maxY),type="o", col="blue", xlab="time", ylab="val") # 状態
par(new=TRUE)
plot(df_all1$time, df_all1$y, xlim=c(0,30), ylim=c(minY,maxY), type="o", xlab="", ylab="") # 観測値
par(new=TRUE)
plot(df_all1$time, df_all1$a, xlim=c(0,30), ylim=c(minY,maxY), type="l", col="green", xlab="", ylab="") # 状態予測
par(new=TRUE)
plot(df_all1$time, ci_a[,1], xlim=c(0,30), ylim=c(minY,maxY), type="l", lty=2, col="green", xlab="", ylab="")
par(new=TRUE)
plot(df_all1$time, ci_a[,2], xlim=c(0,30), ylim=c(minY,maxY), type="l", lty=2, col="green", xlab="", ylab="")
par(new=TRUE)
plot(df_all1$time, df_all1$f, xlim=c(0,30), ylim=c(minY,maxY), type="l", col="red", xlab="", ylab="") # 観測値予測
par(new=TRUE)
plot(df_all1$time, ci_f[,1], xlim=c(0,30), ylim=c(minY,maxY), type="l", lty=2, col="red", xlab="", ylab="")
par(new=TRUE)
plot(df_all1$time, ci_f[,2], xlim=c(0,30), ylim=c(minY,maxY), type="l", lty=2, col="red", xlab="", ylab="")

青(点と線):θ、黒(点と線):Y
緑:状態予測、赤:観測値予測
緑(破線):状態予測の95%信頼区間、赤(破線):観測値予測の95%信頼区間
ローカルレベルモデルの場合は、状態予測値と観測値予測値は同じとなるので、図では緑が赤で上書きされている。
もちろん、信頼区間は異なる。ただし単に観測値誤差(V=3)を足し引きしただけ。
観測値予測の信頼区間は予測区間(time>=21)からのみ。
では、最後にパラメータの推定を考える。
これまでは、モデルのパラメータは与えられたものとして見てきた。
通常のモデリングプロセスでは、パラメータを推定し、カルマンフィルタを実行し状態や予測値の推定をする流れとなるだろう。
ローカルレベルモデルの場合は、V、Wが推定するパラメータとなる。
推定するパラメータを引数とする関数を作成する。
その関数に対し、dlmMLE(データ, パラメータ初期値, 関数)を適用する。dlmMLE()は後ろでoptim()が動いている。
使い方は、関数を定義し、optim()を適用という流れと同じ。
前回記事参照。
関数ではdV=exp(V_estim)のように、expを取った。こうすると、最適値計算において負の値を探索しないらしい。
返り値はV_estim=log(aV)となるので、推定したパラメータの対数が返ってくる。
これからカルマンフィルタを実行するモデルにこれら推定したパラメータを代入する際は、expをとっておく。
まず、dlmMLE()を適用する関数を定義。
fn_mod1 <- function(parm) {
V_estim <- parm[1]
W_estim <- parm[2]
m.0 <- 10 # 状態の初期平均を仮設定
C.0 <- 50 # 状態の初期標準偏差を仮設定
# return( dlm( m0=m.0, C0=C.0, FF=1, V=V_estim, GG=1, W=W_estim ) ) # dlm()を使うとエラーが出る?!?
return( dlmModPoly( order=1, m0=m.0, C0=C.0, dV=exp(V_estim), dW=exp(W_estim) ) )
}
dlm()を使うと推定の際になぜかエラーが出る。原因は不明。なので、dlmModPoly()で定義しておく。
parm_start <- c( 10*runif(1), 10*runif(1) ) # 推定するパラメータ(V、W)の初期値を適当に設定
res2 <- dlmMLE(y=df1$y[-1], parm=parm_start, build=fn_mod1)
V_est <- exp(res2$par[1])
7.681681
W_est <- exp(res2$par[2])
2.406207
V=7.681681、W=2.406207と推定されたが、推定結果は真の値であるV=3、W=6と異なる。
データ数が20で少ないのかと思い、データ数を増やしたりしていろいろとシミュレーションしてみた。数百データがあるとだいたい真の値に近づく傾向があるようである。
次回以降、さまざまな時系列モデルを状態空間表現で定義し、この流れを見て行く。
続く。
